30 de março de 2021
Marissa G. Schamne
PhD | CientistaPesquisadora e professora universitária, farmacêutica por formação. Despertou o interesse pela pesquisa durante a faculdade na Iniciação Científica, que obviamente foi na área da neuropsicofarmacologia. Concluída a graduação, segue o caminho da carreira acadêmica e pesquisa até a co-fundação do Rigor Científico – onde encontrou uma maneira de expressar todo seu amor pela Ciência, e vislumbra a possibilidade de levar essas informações ao maior número possível de pessoas. Era uma pessoa tímida que conforme foi se inserindo no meio acadêmico encontrou uma forma de libertar-se dessa timidez, e agora matraqueia sem parar. Apaixonada pela natureza, esportes ao ar livre e a sensação de liberdade que isso traz. Gosta de viajar pelo mundo, seja através dos livros que lê ou das viagens que faz. Almeja alçar vôos mais altos divulgando ciência por aí.



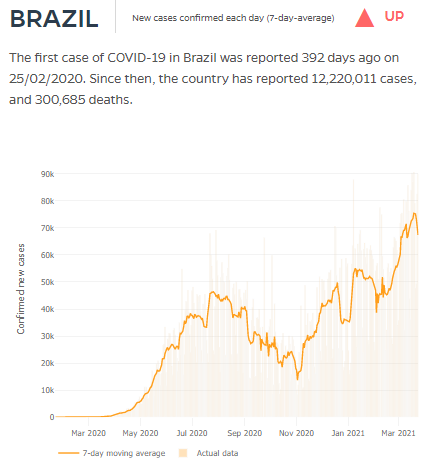
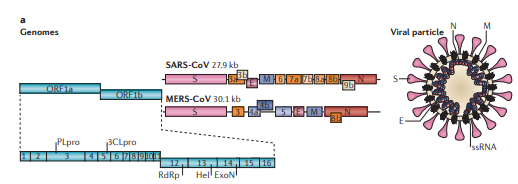
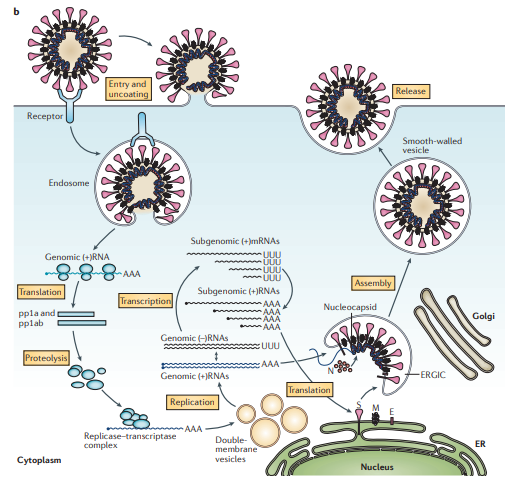
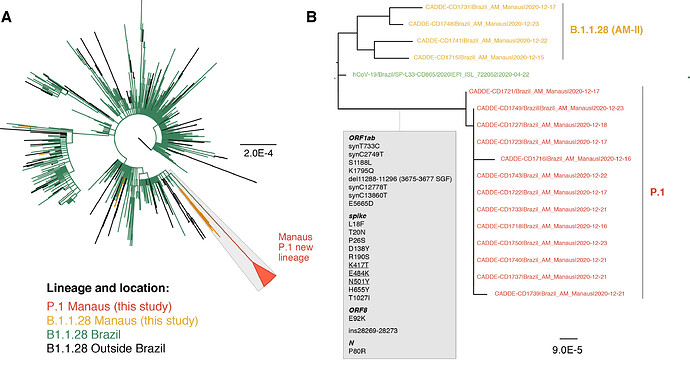



Comentários